Il fattore trascrizionale Zeb1 (Zinc finger E-box binding homebox 1) promuove l’invasione e l’aggressività tumorale inducendo la transizione epitelio-mesenchimale (EMT) nelle cellule tumorali (1-4).
Zhu e colleghi, analizzando in vitro quali fattori trascrizionali si attivassero durante il differenziamento degli osteoclasti (OC), hanno riscontrato che tali cellule, esprimono Zeb1, che era stato finora associato solo alla EMT (2, 3). Per comprenderne meglio il ruolo negli OC, gli autori hanno generato un modello murino knockout condizionale (Zeb1-/-), sopprimendo Zeb1 nei progenitori mieloidi. L’analisi istomorfometrica di animali Zeb1-/- a 3 mesi ha mostrato un fenotipo osteoporotico, con riduzione della BV/TV, Tb.Th. e Tb.N. Inoltre, nonostante non si riscontrassero alterazioni del numero di osteoclasti sulla superficie ossea degli animali Zeb1-/-, gli autori hanno riscontrato un incremento della superficie erosa associata ad elevati livelli sierici di CTX. Nessuna modulazione dei parametri osteoblastici è stata riscontrata.
Esperimenti in vitro hanno dimostrato come le cellule isolate dal midollo degli animali Zeb1-/- non presentano alterazioni della loro capacità differenziativa in OC e dell’espressione dei marker tipici di tali cellule, quali Catepsina K e Fosfatasi Acida Tartrato Resistente. Tuttavia, gli osteoclasti Zeb1-/- erano caratterizzati da una maggiore attività di riassorbimento osseo in quanto formavano lacune di riassorbimento di volume maggiore rispetto a quelle di OC wild type.
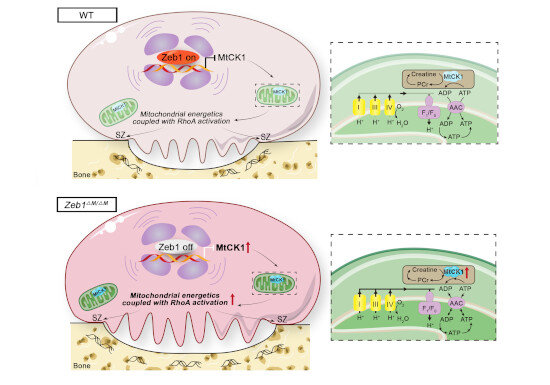
Per comprendere quale fosse il meccanismo molecolare tramite cui Zeb1 regola l’attività degli OC, gli autori hanno effettuato un’analisi trascrittomica ad ampio spettro. Tra i geni più up-regolati negli osteoclasti Zeb1-/- è stato individuato ckmt1, che codifica per l’enzima MtCK1 fondamentale per l’attività ossidativa dei mitocondri e per il metabolismo energetico (5). L’overespressione di MtCK1 in OC WT determina un fenotipo simile a quello degli OC Zeb1-/-, aumentando la respirazione mitocondriale, la produzione di ATP e stimolando l’attività di riassorbimento osseo degli OC. Questi dati sono stati confermati anche in osteoclasti umani, in cui la riduzione specifica dei geni ZEB1 o CKMT1 con delle piccole sequenze nucleotidiche (small interfering RNA), determina un aumento di MtCK1 e dell’attività di riassorbimento osseo.
In questo lavoro, per la prima volta, si associa ZEB1 al metabolismo energetico e si identifica MtCK1 come target a valle di questo fattore di trascrizione, che è espresso in modo ubiquitario ed è coinvolto nella fine regolazione di meccanismi fondamentali per il benessere cellulare. Tale studio dimostra come MtCK1 modulando l’attività di ZEB1, potrebbe rappresentare un bersaglio per nuove strategie terapeutiche.
Commento a “A Zeb1/MtCK1 metabolic axis controls osteoclast activation and skeletal remodeling” di Zhu L, Tang Y, Li XY, Kerk SA, Lyssiotis CA, Feng W, Sun X, Hespe GE, Wang Z, Stemmler MP, Brabletz S, Brabletz T, Keller ET, Ma J, Cho JS, Yang J, Weiss SJ. EMBO Journal (2023) Apr 3;42(7):e111148. doi: 10.15252/embj.2022111148.
Referenze
1. Wu HT, Zhong HT, Li GW, Shen JX, Ye QQ, Zhang ML, et al. Oncogenic functions of the EMT-related transcription factor ZEB1 in breast cancer. J Transl Med. 2020;18(1):51.
2. Perez-Oquendo M, Gibbons DL. Regulation of ZEB1 Function and Molecular Associations in Tumor Progression and Metastasis. Cancers (Basel). 2022;14(8).
3. Zhang P, Sun Y, Ma L. ZEB1: at the crossroads of epithelial-mesenchymal transition, metastasis and therapy resistance. Cell Cycle. 2015;14(4):481-7.
4. Cheng L, Zhou MY, Gu YJ, Chen L, Wang Y. ZEB1: New advances in fibrosis and cancer. Mol Cell Biochem. 2021;476(4):1643-50.
5. Yang M, Wang X, Ye Z, Liu T, Meng Y, Duan Y, et al. Mitochondrial creatine kinase 1 regulates the cell cycle in non-small cell lung cancer via activation of cyclin-dependent kinase 4. Respir Res. 2023;24(1):111.